تحلیل سیگنالهای MEG/EEG با کتابخانه MNE پایتون
- دسته:اخبار علمی
- هما کاشفی
در این مقاله به تحلیل سیگنالهای EEG/MEG با استفاده از پکیج MNE-Python میپردازیم. اینکه چطور دیتاست را بخوانیم، ترایالها را جدا کنیم و یا آنها را رسم کنیم. در این مقاله مهمترین ساختار دادههای پکیج MNE-Python معرفی میشوند: Raw, Epochs.
برای شروع کار، ابتدا نیاز است که ماژولهای ضروری پایتون را import کنیم:
Python
import numpy as np
import mne
خواندن داده با کمک کتابخانه MNE
ساختار دادههای MNE-Python مبتنی بر فرمت فایل fif هستند اما توابعی در این پکیج وجود دارند که با استفاده از آنها میتوان انواع دیگر فرمتهای داده را خواند مانند .gdf همچنین MNE-Python واسطی دارد که با استفاده از آن میتوانید دیتاستهای public را در اختیار داشته باشید. به عبارت دیگر این پکیج MNE، دیتاست های در دسترس عموم را برای شما دانلود و مدیریت میکند. علاوه بر آن میتوانید دیتاستهای خودتان را نیز در این پکیج پردازش کنید.
در این آموزش میخواهیم یکی از دیتاستهای موجود در پکیج MNE را بخوانیم (دیتاستی به نام Sample) که حاوی دادههای EEG و MEG از یک سابجکت است که آزمایش شنوایی-بینایی انجام داده است و به طور همزمان اسکنهای MRI این سابجکت ثبت شده است. تابع mne.datasets.sample.data_path به طور خودکار دیتاست را دانلود میکند و سپس مسیر دایرکتوری دیتاست را بر میگرداند. برای آموزش ما از نسخهی downsample شده و فیلترگذاری شدهی داده استفاده کردهایم Sample_audvis_filt-0-40_raw.fif.
Python
sample_data_folder = mne.datasets.sample.data_path() sample_data_raw_file = (sample_data_folder / "MEG" / "sample" /"sample_audvis_filt-0-40_raw.fif") raw = mne.io.read_raw_fif(sample_data_raw_file) Output:
Opening raw data file /home/circleci/mne_data/MNE-sample-data/MEG/sample/sample_audvis_filt-0-40_raw.fif... Read a total of 4 projection items: PCA-v1 (1 x 102) idle PCA-v2 (1 x 102) idle PCA-v3 (1 x 102) idle Average EEG reference (1 x 60) idle Range : 6450 ... 48149 = 42.956 ... 320.665 secs Ready.
به طور پیش فرض، تابع read_raw_fif برخی اطلاعات مربوط به فایلی که در حال لود شدن است را نشان میدهد: برای مثال به ما میگوید که همراه با دادهی ثبت شده، آیتمهای projection نیز وجود دارند. علاوه بر اطلاعاتی که در قسمت خروجی نشان داده شدهاند، شما میتوانید جزئیات بیشتری از Raw را بدست آورید و تنها کافی است از دستورات زیر استفاده کنید:
Python
print(raw) print(raw.info) Output: <Raw | sample_audvis_filt-0-40_raw.fif, 376 x 41700 (277.7 s), ~3.3 MB, data not loaded> <Info | 15 non-empty values bads: 2 items (MEG 2443, EEG 053) ch_names: MEG 0113, MEG 0112, MEG 0111, MEG 0122, MEG 0123, MEG 0121, MEG ... chs: 204 Gradiometers, 102 Magnetometers, 9 Stimulus, 60 EEG, 1 EOG custom_ref_applied: False dev_head_t: MEG device -> head transform dig: 146 items (3 Cardinal, 4 HPI, 61 EEG, 78 Extra)
file_id: 4 items (dict) highpass: 0.1 Hz hpi_meas: 1 item (list) hpi_results: 1 item (list) lowpass: 40.0 Hz meas_date: 2002-12-03 19:01:10 UTC meas_id: 4 items (dict) nchan: 376 projs: PCA-v1: off, PCA-v2: off, PCA-v3: off, Average EEG reference: off sfreq: 150.2 Hz >
آبجکت Raw همچنین چندین تابع رسم built-in دارد؛ در این بخش ما Power Spectral Density (PSD) هر سنسور را با استفاده از تابع compute_psd محاسبه میکنیم و همچنین هر سنسور را با استفاده از دستور plot رسم میکنیم. تنها فرکانسهای پایینتر از 50 هرتز را رسم میکنیم.
Python
raw.compute_psd(fmax=50).plot(picks="data", exclude="bads") raw.plot(duration=5, n_channels=30)
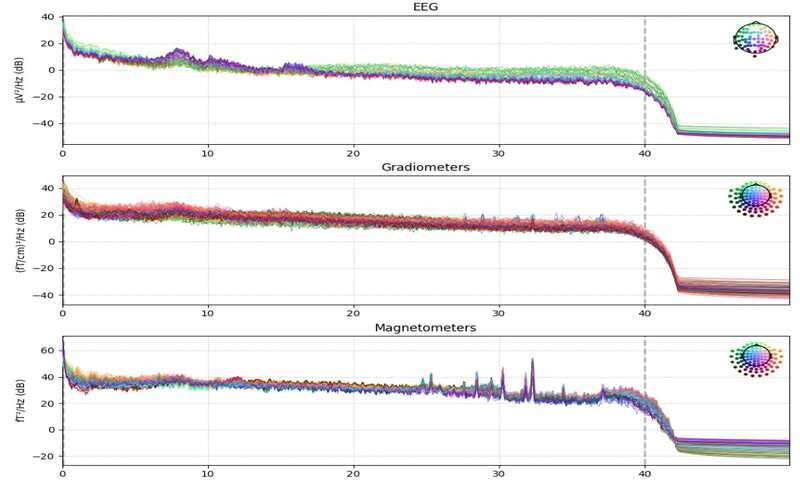
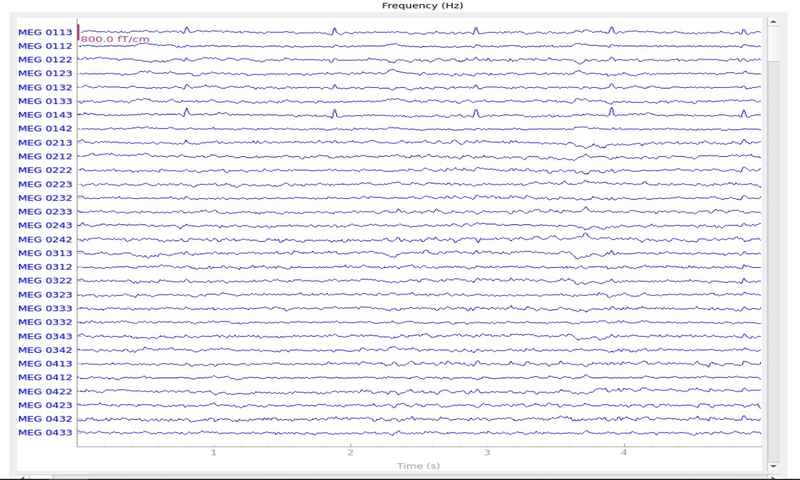
پیش پردازش سیگنال مغزی با کتابخانه MNE
پکیج MNE-Python انواع رویکردها و تکنیکهای پیش پردازش را پشتیبانی میکند (از جمله maxwell filtering، Signal-space projection، independent component analysis، Filtering و Downsampling و …). لیست کامل این رویکردها را میتوانید در ماژولهای mne.preprocessing و mne.filter مشاهده کنید. در این بخش میخواهیم با استفاده از روش ICA داده را تمیز کنیم.
Python
# set up and fit the ICA ica = mne.preprocessing.ICA(n_components=20, random_state=97, max_iter=800) ica.fit(raw) ica.exclude = [1, 2] # details on how we picked these are omitted here ica.plot_properties(raw, picks=ica.exclude)
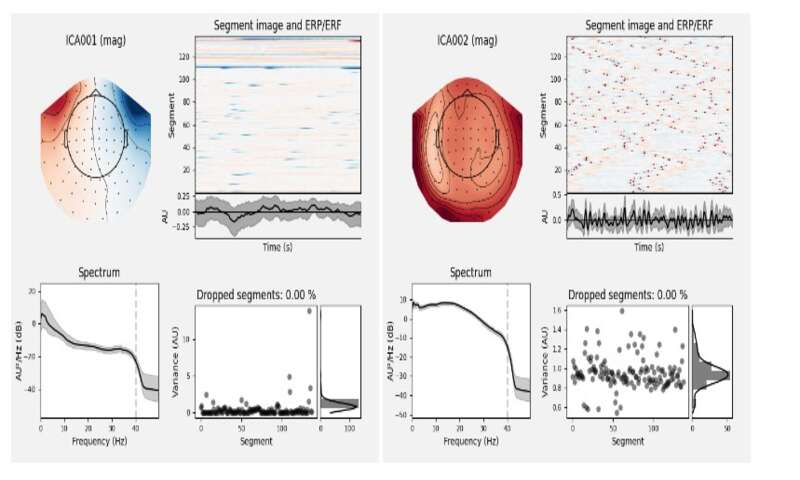
بعد از آنکه مطمئن شدیم کدامیک از کامپوننتها را میخواهیم حذف کنیم آنها را به پارامتر exclude میدهیم سپس ICA را روی سیگنال خام اعمال میکنیم. متد apply مستلزم آن است که دادهی خام در حافظه بارگذاری شود برای این منظور ابتدا از load_data استفاده میکنیم.
جدا کردن ترایالها از دادهی پیوسته با کمک کتابخانه MNE
آبجکت Raw و آرایههای رویداد نیاز هستند تا بتوان آبجکت Epochs را ساخت. Epochs در MNE همان ترایال در واسط مغز کامپیوتر است. برای ساخت آن کافی است از کلاس Epochs استفاده کنیم. باید اطلاعات مربوط به ترایالها را با پارامتر event_id به تابع mne.Epochs بدهیم و tmin و tmax (ثانیه ی ابتدا و انتهای ترایال) را مشخص کنیم. به طور پیش فرض دادههای Raw و Epochs در حافظه قرار نمیگیرند (آنها روی دیسک قرار دارند. اما اینجا با استفاده از پارامتر preload=True، این دادهها را روی حافظه قرار میدهیم.
Python
epochs = mne.Epochs(raw, events, event_id=event_dict, tmin=-0.2,tmax=0.5, reject=reject_criteria,preload=True,) Output: Not setting metadata 319 matching events found Setting baseline interval to [-0.19979521315838786, 0.0] s Applying baseline correction (mode: mean) Created an SSP operator (subspace dimension = 4) 4 projection items activated Using data from preloaded Raw for 319 events and 106 original time points ... Rejecting epoch based on EOG : ['EOG 061'] Rejecting epoch based on EOG : ['EOG 061'] Rejecting epoch based on MAG : ['MEG 1711'] Rejecting epoch based on EOG : ['EOG 061'] Rejecting epoch based on EOG : ['EOG 061'] Rejecting epoch based on MAG : ['MEG 1711'] Rejecting epoch based on EEG : ['EEG 008'] Rejecting epoch based on EOG : ['EOG 061'] Rejecting epoch based on EOG : ['EOG 061'] Rejecting epoch based on EOG : ['EOG 061'] 10 bad epochs dropped
در دورهی پکیج MNE پایتون که به زودی آماده خواهد شد، به تمامی این موارد با جزئیات بیشتری میپردازیم.
با ما همراه باشید…


دیدگاه ها